1.はじめに:なぜ今、GSEAが臨床研究で重要なのか
現代の医療研究、特にがん治療や難病解明の分野では、RNA-seqなどの次世代シーケンシング技術を用いた網羅的な遺伝子発現解析が欠かせません。しかし、膨大なデータから「どの分子が真のターゲットなのか」を見極めるのは容易ではありません。そこで威力を発揮するのが、今回解説するGSEA(Gene Set Enrichment Analysis:遺伝子セット濃縮解析)です。
GSEAは、個々の遺伝子だけを見るのではなく、関連する機能を持つ「遺伝子の集まり」として解析を行います。これにより、単なる数値の羅列を超えて、患者さんの体内で「どのような生物学的プロセス(代謝やシグナル伝達など)が動いているか」を直感的に理解できるようになります。本記事では、GSEAの基礎から実践、結果の解釈までを、医療現場の視点で分かりやすく紐解いていきます。
2.GSEAの基本コンセプト — 「個」ではなく「群」を見る
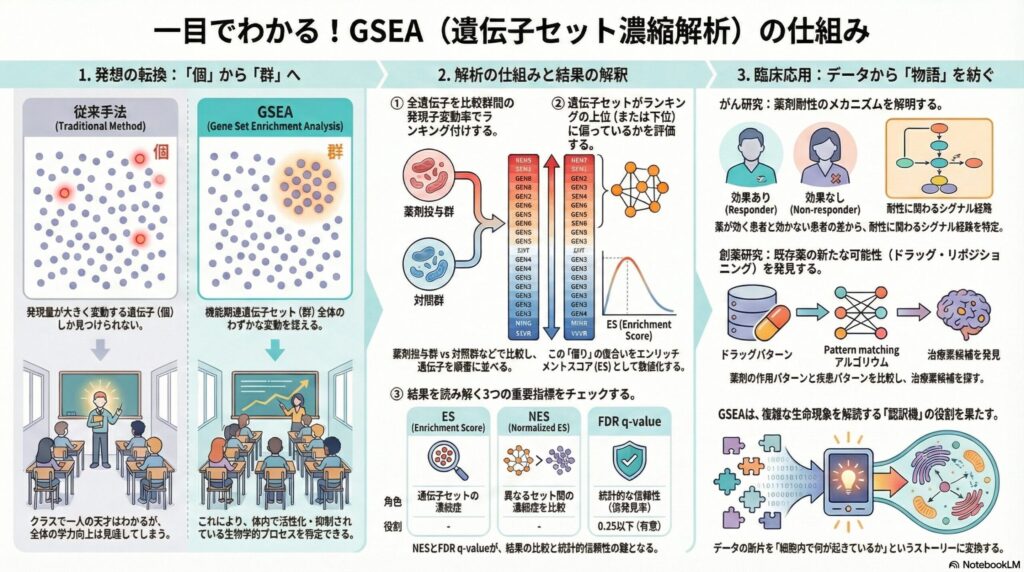
従来の解析手法(差次発現解析)は、いわば「クラスの中でテストの点が極端に高い生徒だけを探す」作業でした。しかし、これでは「平均点は少しずつ上がっているが、クラス全体の学力が向上している兆し」を見落としてしまいます。GSEAは、クラス(遺伝子セット)全体としての傾向を評価する手法です。
具体的には、特定の生物学的機能(例:炎症反応、細胞周期、薬剤代謝)に関連することが既に分かっている遺伝子のリストを「遺伝子セット」として定義します。このセットに含まれる遺伝子たちが、解析対象のデータの中で上位(高発現側)または下位(低発現側)に「濃縮」しているかどうかを統計的に判定するのがGSEAの役割です。
このアプローチの強みは、統計的な有意差(p値)のカットオフにかからない程度の微細な変動であっても、それが一連の経路で共通して起きていれば検出できる点にあります。臨床データのような個人差が激しいサンプルにおいて、この特性は非常に強力な武器となります。
3.解析の心臓部 — ランキングとエンリッチメントスコア
GSEAの解析プロセスで最も重要なのは、全遺伝子を特定の基準で「並び替える(ランキングする)」ことです。一般的には、薬剤投与群と対照群を比較した際の、発現量の変化率(log2 Fold Change)や統計的有意性(p値)を組み合わせてスコア化し、高い順から低い順へと遺伝子を並べます。
次に、あらかじめ用意した「遺伝子セット」のメンバーが、このランキングのどこに位置しているかを確認します。もし、セット内の遺伝子たちがランキングの上位に固まっていれば「その機能は活性化している」と判断できます。この偏り具合を数値化したものが、エンリッチメントスコア(ES)です。
ESの計算には「ランダムウォーク」という手法が使われます。ランキングを上から順に見ていき、遺伝子セットに含まれる遺伝子があればスコアを加点し、含まれなければ減点します。このスコアの最大値(または最小値)がESとなります。この数値が高いほど、その機能が強く関与していることを示唆します。
4.結果を読み解くための3つの重要指標(ES, NES, FDR)
GSEAの結果画面には、複数の統計量が表示されます。医療論文を読み解く際や、自ら解析を行う際に必ずチェックすべき指標は、以下の3点に集約されます。
- ES(Enrichment Score): 遺伝子セットの濃縮度合い。ただし、セットに含まれる遺伝子数によって値が左右されるため、これだけで比較はできません。
- NES(Normalized Enrichment Score): ESを遺伝子セットのサイズで補正したもの。異なるセット間で「どちらの方がより強く濃縮しているか」を比較する際には、このNESを用います。
- FDR q-value(偽発見率): 多くの遺伝子セットを同時にテストするため、偶然に有意な結果が出る確率を補正した値です。一般に0.25以下、より厳密には0.05以下を有意と判断します。
特に「エンリッチメントプロット」と呼ばれるグラフは重要です。緑色の曲線が大きく盛り上がっている部分は、その遺伝子セットが強く濃縮していることを視覚的に示しており、論文での図表説明(Figure Legend)のメインとなります。
5.実践的な解析手順 — データの準備からツール実行まで
実際にGSEAを行う際の手順は、大きく分けて以下の4つのステップとなります。専門的な知識が必要な部分もありますが、最近ではR言語のパッケージ(clusterProfilerなど)や、GUIベースのデスクトップアプリ(GSEA Desktop App)が普及しており、以前よりハードルは下がっています。
- 発現データの整理: RNA-seqなどで得られたカウントデータを正規化し、比較群ごとの発現差異を計算します。
- 遺伝子IDの統一: 解析で使用するデータベース(MSigDBなど)に合わせて、遺伝子名を「Entrez ID」や「Gene Symbol」に変換します。この変換が不正確だと、解析精度が著しく低下するため注意が必要です。
- データベースの選択: どの「遺伝子セット」を使って解析するかを決めます。代表的なものに、代謝経路を集めた「KEGG」や、生物学的プロセスを分類した「GO(Gene Ontology)」、がん研究に特化した「Hallmark」などがあります。
- 解析の実行と可視化: パラメータ(ランダムサンプリングの回数など)を設定して計算を実行します。結果はドットプロットやリッジプロットとして可視化し、直感的に解釈できるように整えます。
6.臨床研究・創薬研究におけるGSEAの応用事例
GSEAは、すでに多くの臨床研究でその価値が証明されています。例えばがん研究においては、特定の分子標的薬に対して「感受性がある患者群」と「耐性がある患者群」の生検サンプルを比較することで、耐性に関与するシグナル伝達経路を特定するのに役立っています。
また、ドラッグ・リポジショニング(既存薬の転用)の研究においても、GSEAは有用です。ある薬剤を投与した際の遺伝子変動パターン(シグネチャー)が、特定の疾患の改善パターンと逆転していれば、その薬剤が治療薬候補になる可能性があるからです。
このように、GSEAは「どの遺伝子が変わったか」という情報の断片を、「細胞内で何が起きているか」というストーリーに紡ぎ直す役割を担っています。これにより、研究者は次の実験の仮説を立てやすくなり、医師は病態の背景にある分子メカニズムをより深く考察することが可能になるのです。
7.おわりに:GSEAが切り拓く精密医療の未来
GSEAは、複雑な生命現象をデジタルデータから解釈するための、なくてはならない「翻訳機」のような存在です。単一のバイオマーカー探しに留まらず、ネットワークとしての生命の動きを捉えるこの手法は、まさに一人ひとりに最適な治療を提供する「精密医療(プレシジョン・メディシン)」の基盤を支えています。
バイオインフォマティクスは日々進化していますが、GSEAの持つ「機能単位での解釈」という哲学は、今後も変わることなく重要視され続けるでしょう。本記事が、皆様の臨床研究やデータ解析の一助となれば幸いです。
免責事項
本記事の内容は、公開時点での信頼できる情報に基づき作成されていますが、解析結果の正確性や特定の治療効果を保証するものではありません。実際の解析や臨床判断においては、最新のガイドラインや専門家の指示に従ってください。本記事の利用により生じたいかなる損害についても、当ラボは一切の責任を負わないものとします。
本記事は生成AI (Gemini) を活用して作成しています。内容については十分に精査しておりますが、誤りが含まれる可能性があります。お気づきの点がございましたら、コメントにてご指摘いただけますと幸いです。
Amazonでこの関連書籍「改訂 独習Pythonバイオ情報解析〜生成AI時代に活きるJupyter、NumPy、pandas、Matplotlib、Scanpyの基礎を身につけ、シングルセル、RNA-Seqデータ解析を自分の手で (実験医学別冊) 」を見る