1.はじめに:AI創薬における「質の高いデータ」の重要性
現代の創薬シーンにおいて、コンピュータを用いた「AI創薬」や「イン・シリコ(in silico)スクリーニング」は欠かせない存在となっています。膨大な化合物ライブラリから、標的となるタンパク質に結合する「鍵」を見つけ出す作業は、創薬の初期段階を大幅に加速させます。
しかし、シミュレーションの結果が現実の生物学的反応を反映するためには、解析の土台となる「タンパク質構造データ」が正確でなければなりません。ITの世界には「Garbage In, Garbage Out(ゴミを入れればゴミが出る)」という言葉がありますが、創薬計算においても全く同じことが言えます。
本記事では、世界中で広く利用されている構造解析ソフト「UCSF Chimera(チメラ)」を用い、ドッキングシミュレーションの前段階で行うべき「タンパク質準備(プロテイン・プレパレーション)」の全手順を、医療関係者の方々に向けて分かりやすく解説します。
2.UCSF Chimeraとは:分子構造を可視化・操作する強力なツール
UCSF Chimeraは、カリフォルニア大学サンフランシスコ校(UCSF)で開発された、生体分子構造を3次元で表示・分析するためのソフトウェアです。タンパク質や核酸(DNA/RNA)などの複雑な構造を、マウス操作で直感的に観察できるのが大きな特徴です。
創薬研究においては、標的タンパク質と治療薬候補(リガンド)がどのように結合するかを予測する「ドッキングシミュレーション」の準備作業に頻繁に活用されます。このソフトを使うことで、実験で得られた生の構造データを、計算機が正しく認識できる形へと整えることができます。
なお、現在は後継版である「UCSF ChimeraX」の開発が進んでおり、より高度な描画や解析が可能になっています。しかし、長年蓄積されたチュートリアルや既存のツールとの互換性の観点から、従来のChimeraも依然として多くの研究現場で現役として使用されています。
3.なぜドッキング前に「タンパク質の準備」が必要なのか
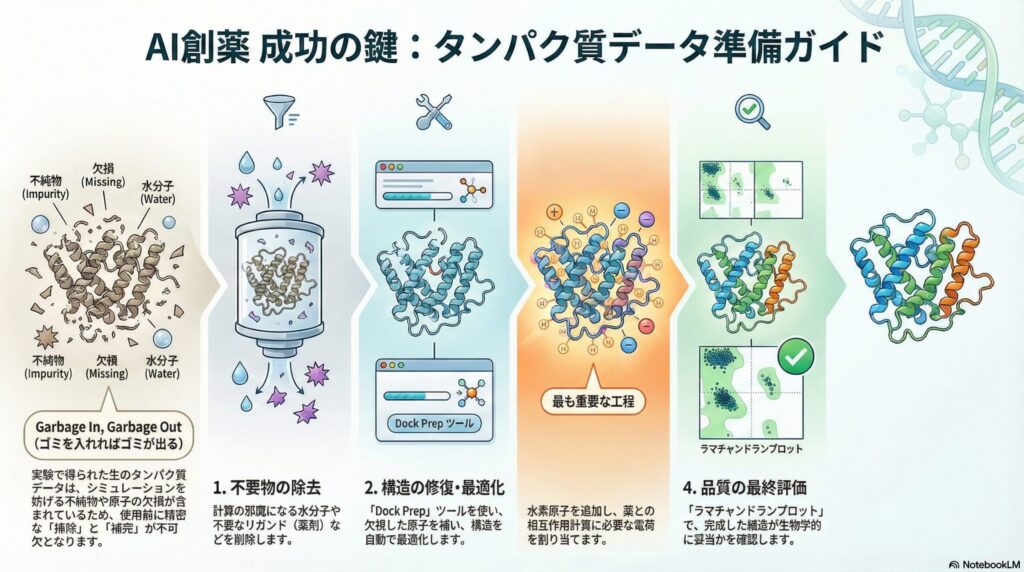
実験手法(X線結晶構造解析など)によって決定されたタンパク質の構造データ(PDBファイル)は、そのままではシミュレーションに使用できないことがほとんどです。なぜなら、これらのデータには計算を妨げる不純物や、データ上の「欠落」が含まれているからです。
具体的には、結晶化の際に使用された「水分子」や「不要なイオン」が結合部位を塞いでいたり、本来存在するはずの「水素原子」の情報が抜け落ちていたりします。水素原子は原子量が小さいため、X線解析では捉えにくく、データから欠落しやすいのです。
これらの問題を放置したままドッキングを行うと、化合物がタンパク質に正しく入り込めなかったり、静電気的な相互作用(電荷の引き合い)を正しく計算できなかったりします。そのため、構造の「掃除」と「補完」を行う準備プロセスが、解析の成否を分ける鍵となります。
4.ステップ別:タンパク質構造の最適化手順
ステップ1:ターゲット構造の取得と初期確認
まずは、解析対象となるタンパク質のデータを取得します。世界的なデータベースであるPDB(Protein Data Bank)から、特定のID(4桁の英数字)を用いて直接ソフト内に読み込むことが可能です。
読み込まれた構造を眺め、タンパク質のどこに活性部位(薬が結合する場所)があるか、全体像を把握します。この際、タンパク質の表面に異常な突起がないか、あるいは構造の一部が不自然に途切れていないかを視覚的に確認することが、最初の大切な一歩となります。
ステップ2:不要な分子(水・配位子)の除去
結晶構造には、タンパク質の周囲を取り囲む水分子が含まれています。これらは実際の体内では流動的ですが、結晶データ上は固定されています。ドッキングの邪魔になることが多いため、基本的にはこれらの水分子を選択して削除します。
また、以前の研究で使用された共結晶の薬剤(リガンド)や、実験用のバッファー成分(イオンなど)も残っている場合があります。これらが残っていると、新しい化合物が結合するスペースがなくなってしまうため、ターゲットとなるタンパク質本体のみを残すようにクリーンアップを行います。
ステップ3:Dock Prepツールによる自動最適化
Chimeraには「Dock Prep」という非常に便利な統合ツールが備わっています。これを使うことで、複雑な前処理を半自動で行うことができます。Dock Prepは、不完全な側鎖(アミノ酸の枝分かれ部分)の修復や、欠損している原子の補完を提案してくれます。
このプロセスでは、アミノ酸の立体的な配置を最適化するために「Dunbrackロタマーライブラリ」などの統計的なデータが参照されます。これにより、エネルギー的に安定した、より自然なタンパク質の形へと修正することが可能になります。
ステップ4:水素原子の追加と電荷の割り当て
最も重要なのが、水素原子の追加です。水素は結合の安定性を決める「水素結合」の主役です。Chimeraでは、周囲の環境(pHなど)を考慮して、水素がどの向きに付くべきかを計算し、自動的に追加してくれます。
さらに、原子ごとの電気的な偏りを示す「部分電荷(Partial Charge)」を割り当てます。これにより、タンパク質と薬の間に働くプラスとマイナスの引き合いを、物理学的に正しく計算できるようになります。この設定が正確であるほど、ドッキングの予測精度は向上します。
5.構造の品質評価:ラマチャンドランプロットの活用
準備が整ったタンパク質構造が、生物学的に妥当であるかどうかを評価する必要があります。その代表的な指標が「ラマチャンドランプロット」です。これは、アミノ酸同士の結合角度が「自然な範囲」に収まっているかをグラフ化したものです。
タンパク質を構成するアミノ酸の鎖は、物理的に曲がれる角度が決まっています。無理な角度で固定されている箇所は、構造データとしての信頼性が低いことを意味します。Chimeraの解析ツールを使えば、この角度の異常を瞬時に可視化できます。
理想的な構造では、90%以上の残基が「許容領域」と呼ばれるグラフ上の安定したエリアに配置されます。もし異常に多くの残基が領域外にある場合は、その構造を用いたシミュレーション結果には注意が必要です。構造の再調整や、別のPDBデータの使用を検討すべきサインとなります。
6.トラブルシューティングとベストプラクティス
前処理の過程では、しばしばエラーに遭遇します。例えば、特定の残基(アミノ酸)がひどく損傷していて、自動修復がうまくいかないケースです。その場合は、コマンドラインから手動でアミノ酸を置換(Mutation)したり、削除したりする柔軟な対応が求められます。
また、最良の結果を得るためのコツは、各ステップが終わるごとに構造を「自分の目」で確認することです。自動ツールは強力ですが、万能ではありません。水素原子が不自然な方向を向いていないか、大事な結合部位が削られていないか、専門家としての視点が品質を担保します。
最後に、準備が完了したファイルは、電荷情報を含む「Mol2形式」などで保存します。この際、作業履歴を残しておくことも重要です。どのPDB IDを使い、どのような処理を施したかを記録しておくことで、研究の再現性を確保することができます。
7.まとめ:正確な前処理が創薬の未来を切り拓く
UCSF Chimeraを用いたタンパク質の準備と品質評価は、一見地味で根気のいる作業かもしれません。しかし、この丁寧なプロセスこそが、信頼性の高いドッキング結果を生み出し、ひいては次世代の画期的な新薬創出へと繋がっていきます。
デジタル技術と生命科学が融合するAI創薬の現場において、ソフトウェアを正しく使いこなし、データの質を見極める能力は、今後ますます重要になるでしょう。本記事が、皆様の研究や業務の一助となれば幸いです。
免責事項
本記事の内容は、公開時点での一般的な学術情報に基づき作成されています。個別の研究プロジェクトにおけるソフトウェアの使用結果について、当ラボはいかなる保証もいたしません。ソフトウェアの操作ミスやデータの不備に起因する研究上の損失や損害について、当ラボは一切の責任は負わないものとします。実際の研究に際しては、各ソフトの公式ドキュメントを参照し、自己責任において実行してください。
本記事は生成AI (Gemini) を活用して作成しています。内容については十分に精査しておりますが、誤りが含まれる可能性があります。お気づきの点がございましたら、コメントにてご指摘いただけますと幸いです。
Amazonでこの関連書籍「Pythonではじめるバイオインフォマティクス ―可読性・拡張性・再現性のあるコードを書くために」を見る